作為生命活動的核心元素
RNA具有復雜的三維結構與動態性特征
直接調控著基因表達、病毒復制
等關鍵生物過程
然而RNA結構研究長期存在技術瓶頸
成為科學界的一大難題
今天(3月14日)
復旦大學與四川大學華西醫院團隊的
合作研究成果
以“Cryo-EM reveals mechanisms
of natural RNA multivalency”為題
發表在《科學》(Science)雜志上
作為復旦AI4S(AI for Science)的又一碩果
該研究創新性地將深度學習技術
與冷凍電鏡技術相結合
為RNA生物學研究開辟了全新的技術路徑
不僅顯著提升RNA結構解析的效率
更為新藥研發提供了重要技術支撐和理論指導
1突破RNA結構研究的“盲盒”困境
傳統的RNA結構解析方法面臨實驗成本高昂、預測精度不足等難題。冷凍電鏡雖然能夠解析高分辨率結構,但要篩選出能夠形成穩定構象的RNA序列,仍然需要耗費大量資源。此外,現有算法還依賴于Rfam數據庫等二級結構信息。
“RNA有很多家族,此前在解析RNA結構時,科研人員往往不確定應該選擇哪些序列進行解析。”復旦大學智能復雜體系實驗室研究員、文章共同通訊作者孫思琦解釋道,“這就像開‘盲盒’,我們只能猜測哪些家族的序列可能具有穩定的結構。”
面對這一困境,團隊決定引入人工智能技術。孫思琦本科畢業于復旦大學數學科學學院,博士期間在美國攻讀計算機專業,研究方向為計算生物學,畢業后在美國微軟研究院開展大語言模型相關的研究。回國后,他和團隊開始嘗試用AI大模型解決生物問題,RNA結構研究是他們持續關注的方向之一。復旦大學智能復雜體系實驗室博士生吳浩、許晟為本文共同第一作者。
孫思琦團隊與合作團隊共同突破三大技術:提出高速高靈敏度的蛋白質同源物檢測方法,實現遠程同源物的快速識別;開發高精度端到端RNA三維結構預測方法,建立全鏈條結構解析框架;設計基于非自回歸神經網絡的蛋白質譜快速解析算法,攻克質譜數據高效解碼難題。通過融合大語言模型與對比學習技術,該系列成果在蛋白質檢測、RNA結構預測、蛋白質測序等方向同步提升AI算法的速度與精度。相關研究成果于近期發表于《自然》(Nature)子刊,為本工作奠定了基礎。
在這項最新研究中,復旦大學與華西醫院團隊緊密合作。復旦大學團隊負責AI算法的開發,而四川大學華西醫院則負責使用冷凍電鏡對RNA結構進行解析。
“通過將AI與實驗相結合,我們可以精確預測出哪些RNA序列具有穩定的結構。”孫思琦表示。基于AI的分析結果,實驗人員在解析這些結構時,只需關注那些被推薦的序列,科研效率得到了大幅提升。
2自主研發RNA序列大模型
此前,在RNA結構預測領域,傳統AI方法往往局限于已知結構的簡單解析。聯合團隊創新性地將深度學習與冷凍電鏡結合,研發出CRAFTS智能篩選系統,實現對多聚體RNA復雜構象的高效預測,為解析動態RNA結構提供了“AI導航儀”。
團隊研發的CRAFTS模型(Contrastive RNA learning For sTructure Screening),利用深度學習技術,能夠從海量RNA序列中挖掘潛在的折疊規律,為冷凍電鏡實驗提供精準的篩選工具,從而顯著提高效率并降低實驗成本。
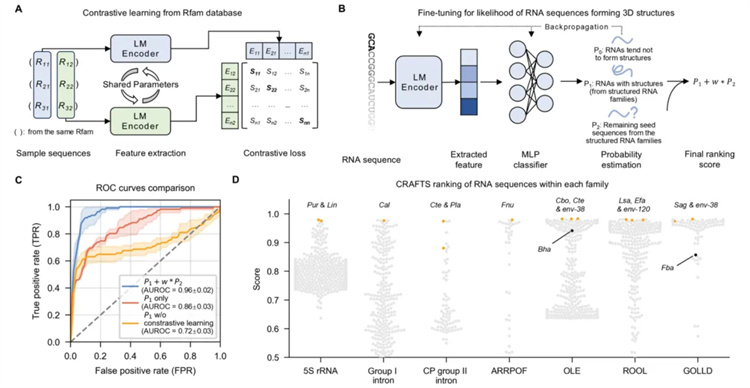
圖2:CRAFTS的性能評估,分析RNA家族結構特征。A. 對比學習用于確定輸入對是否屬于同一RNA家族。 B. 微調模型以從每個RNA家族的序列中提取結構特征。 C基準數據性能表現:平均ROC曲線及標準差(基于5折交叉驗證)。D. 對5S rRNA、I類內含子、CP II類內含子等家族分析。通過實驗結構測定驗證的序列以橙色標注。
在技術架構和創新應用上,CRAFTS模型展現了顯著的突破性進展。基于RNAcentral、NCBI等權威數據庫的10億多條非冗余RNA序列,模型通過自監督學習提取RNA序列的語義特征,生成深度表征。
基于該模型,團隊構建了超過900萬對RNA序列,最大化同一RNA家族內序列的相似性,最小化不同RNA家族序列之間的相似性,從而精準提取家族特異的結構特征。
團隊利用Rfam數據庫中4038個RNA家族的數據進行訓練,整合了未解析的RNA種子序列作為偽負類,顯著擴展了訓練數據的范圍。這種數據增強策略大幅提升了模型的泛化能力,尤其是在數據稀缺的場景下,模型表現尤為突出。在僅有364個正樣本的訓練集中,CRAFTS通過5折交叉驗證展現了高魯棒性,測試結果的標準差低于0.03。
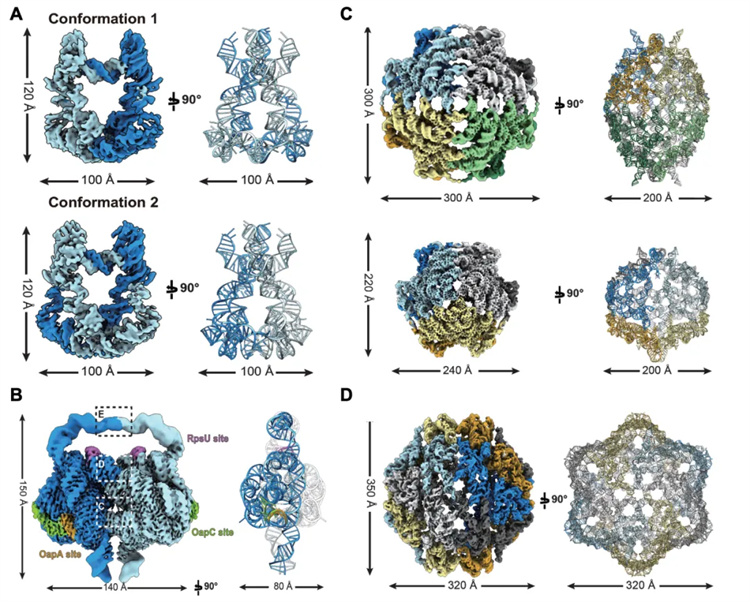
圖3:冷凍電鏡結構。A. ARRPOF 二聚體雙構象冷凍電鏡結構。B. OLE 二聚體冷凍電鏡結構。C. ROOL 六聚體和八聚體冷凍電鏡結構。D. GOLLD 十二聚體冷凍電鏡結構。
實際應用中,CRAFTS模型不僅在經典RNA家族(如5S rRNA、I型內含子和II型內含子)中表現優異,還成功應用于ARRPOF、OLE、ROOL和GOLLD等新RNA家族的結構篩選。
CRAFTS模型與冷凍電鏡技術的深度協同,成功突破了RNA結構篩選中的“盲盒”困境,為RNA結構生物學研究提供了全新的工具。這一研究成果不僅提升了RNA三級結構解析的效率,還為探索RNA多態性在生命活動中的潛在功能開辟了新的可能性。
“RNA的結構預測,其實還遠遠沒有被完全解決。”孫思琦表示,通過進一步優化模型,可以提升其在不同RNA家族中的預測能力。“目前RNA相關的高質量數據非常有限,AI賦能的科研方法將在未來發揮更大的優勢。”
基于RNA結構分析和篩選,科研人員能夠更有效地判斷哪些分子可以與RNA結合,從而加速RNA小分子藥物的研發。這一突破不僅顯著提升了RNA結構解析的精度和效率,更為新藥研發提供了重要的技術支撐和理論指導。